
事后配对比较是数据科学家日常工作中非常常见的一部分。每当我们做方差分析或非参数测试时,有两个以上的组,这是不可避免的。
在某些时候,你可能希望有一个很好的可视化,说明哪些是显著的,哪些是不显著的。可能会在图上写上P值。那么你可以考虑用自己编写的函数来完成这项工作。或者你可以找到一个 ggpubr 包作为简单的解决方案。陷阱来了。作为例子给出的通常的代码并不能产生为多重比较而调整的p值!假设你有一些Y反应和4个组A、B、C、D,你想比较其他组中的Y和A参考组中的Y。那么你最终会得到这样的代码。
library(tidyverse)
library(rstatix)
library(ggpubr)
library(ggplot2)
my_comparisons <- list( c( "A", "B"), c( "A", "C"), c( "A", "D")
ggboxplot(data, x = "分组.变量", y = "你的.反应",
palette = "jco",fill="grouping.variable",order=c("A", "B", "C", "D")。
图例="右")+
stat_compare_means(comparisons = my_comparisons, label.y = c(0.4, 0.5, 0.6),method="wilcox.test", p.adjust.method = "bonferroni")+
labs(y="Y反应",x="这里的一些分组变量",fill="组")
还有那张没有调整过的P值的图!
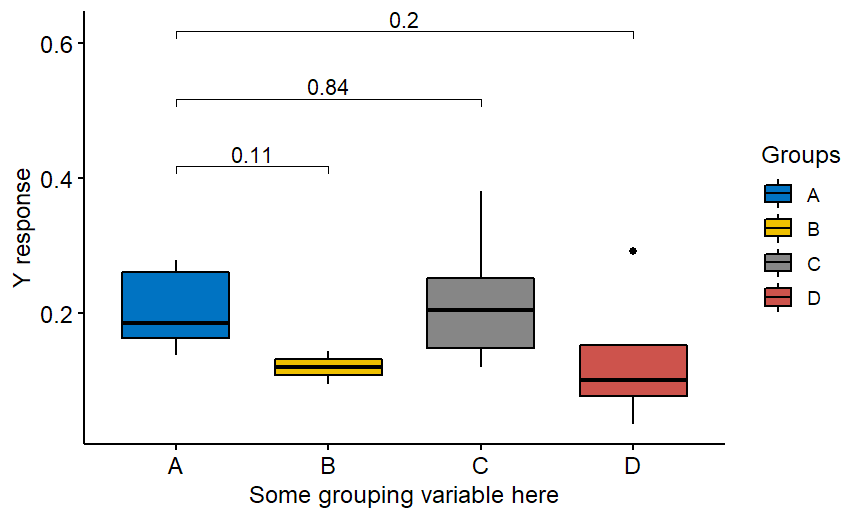
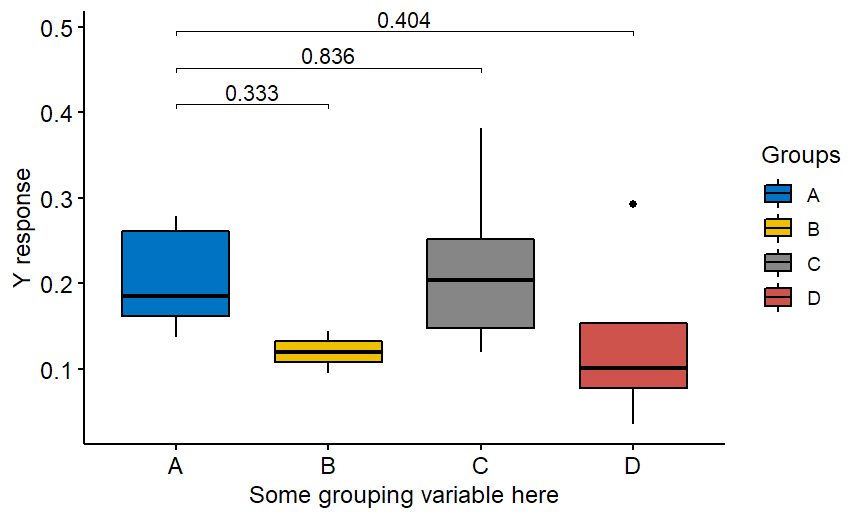
正确的代码如下。
stat.test %
wilcox_test(your.response ~ grouping.variable,ref.group = "A") %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(data, x = "分组变量", y = "你的反应",
palette = "jco",fill="grouping.variable",order=c("A", "B", "C", "D"),
legend="right")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0.01)+
labs(y="Y反应",x="这里的一些分组变量",fill="组")
当然,上面是一个使用Wilcoxon非参数检验的例子,但该软件包也提供t检验。如果你想比较 "一切与一切",而不仅仅是与A组比较,那么只需从代码中删除ref.group = "A"。
stat.test %
wilcox_test(your.response ~ grouping.variable) %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(data, x = "分组变量", y = "你的反应",
palette = "jco",fill="grouping.variable",order=c("A", "B", "C", "D"),
legend="right")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0.01)+
labs(y="Y反应",x="这里的一些分组变量",fill="组")而你会得到。
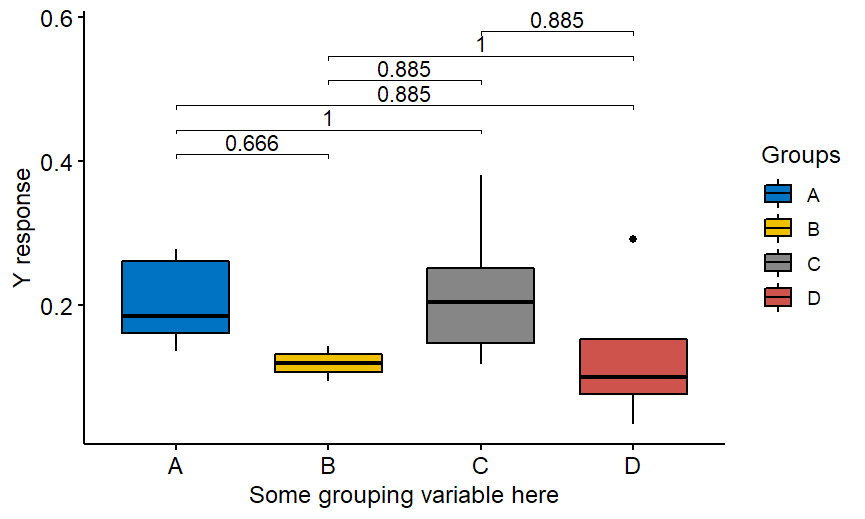
注意,上面的调整方法已经不是Bonferroni了。它是Holm,一个在R中实现的默认方法。 祝你愉快!
