
Porównania post-hoc parami są bardzo częstą częścią codziennej rutyny naukowca. Jest to nieuniknione, gdy przeprowadzamy ANOVA lub testy nieparametryczne z więcej niż dwiema grupami.
W pewnym momencie może pojawić się potrzeba stworzenia ładnej wizualizacji tego, co jest istotne, a co nie. Ewentualnie z wartościami p zapisanymi na wykresie. Można wtedy wymyślić jakąś własną funkcję, która wykona to zadanie. Można też znaleźć ggpubr pakiet jako łatwe rozwiązanie. I tu pojawia się pułapka. Zwykły kod podany jako przykład nie daje wartości p skorygowanych o wielokrotne porównania! Załóżmy, że masz pewną odpowiedź y i 4 grupy A, B, C , D i chcesz porównać y w innych grupach z y w grupie odniesienia A. Wówczas otrzymamy taki kod jak poniżej:
library(tidyverse)
biblioteka(rstatix)
biblioteka(ggpubr)
biblioteka(ggplot2)
my_comparisons <- list( c("A", "B"), c("A", "C"), c("A", "D"))
ggboxplot(dane, x = "grupowanie.zmienna", y = "twoja.odpowiedź",
palette = "jco",fill="grouping.variable",order=c("A", "B", "C", "D"),
legend="right")+
stat_compare_means(comparisons = my_comparisons, label.y = c(0.4, 0.5, 0.6),method="wilcox.test",p.adjust.method = "bonferroni")+
labs(y="Odpowiedź Y",x="Jakaś zmienna grupująca tutaj",fill="Grupy")
I wykres, który NIE ma skorygowanych wartości p!
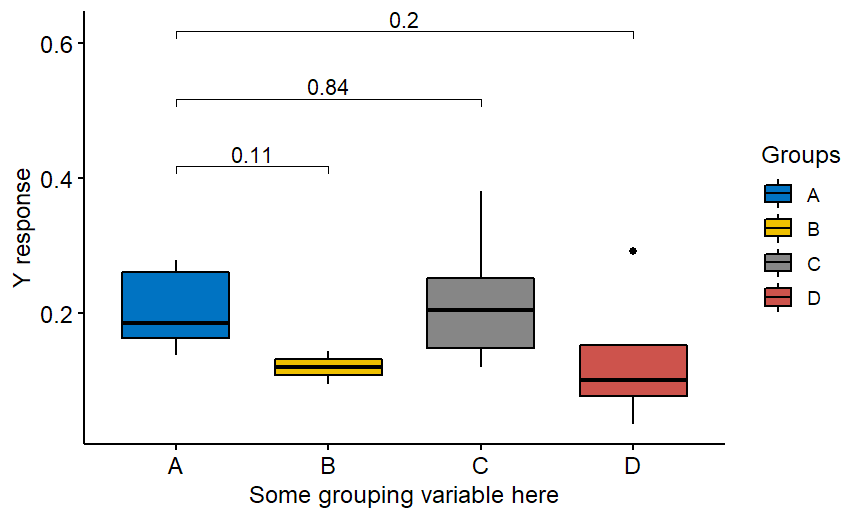
Prawidłowy kod jest następujący:
stat.test %
wilcox_test(your.response ~ grouping.variable,ref.group = "A") %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(data, x = "grouping.variable", y = "your.response",
palette = "jco",fill="grouping.variable",order=c("A", "B", "C", "D"),
legend="right")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0,01)+
labs(y="Odpowiedź Y",x="Jakaś zmienna grupująca tutaj",fill="Grupy")
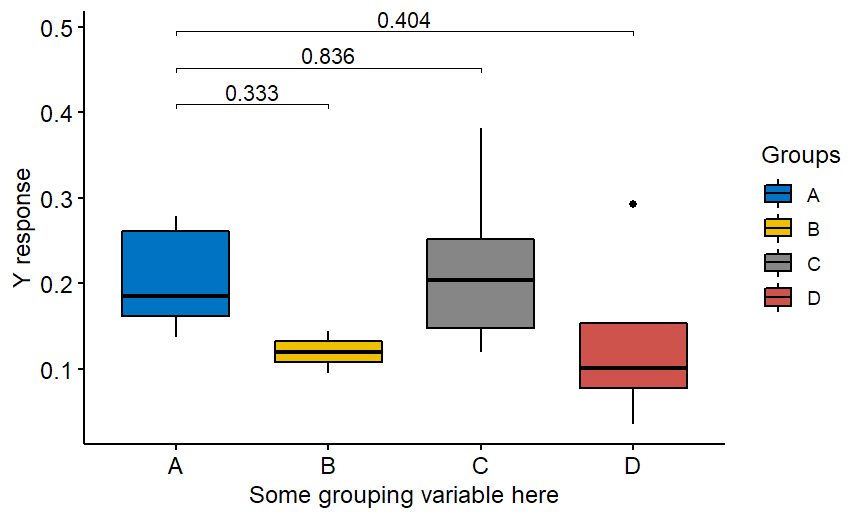
Oczywiście powyżej przedstawiono przykład z testem nieparametrycznym Wilcoxona, ale pakiet oferuje również test t. Jeśli chcesz porównać "wszystko ze wszystkim", a nie tylko z grupą A, po prostu usuń ref.group = "A" z kodu:
stat.test %
wilcox_test(twoja.odpowiedź ~ grupowanie.zmienna) %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(dane, x = "grupowanie.zmienna", y = "twoja.odpowiedź",
palette = "jco",fill="grouping.variable",order=c("A", "B", "C", "D"),
legend="right")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0,01)+
labs(y="Odpowiedź Y",x="Jakaś zmienna grupująca tutaj",fill="Grupy")I otrzymujesz:
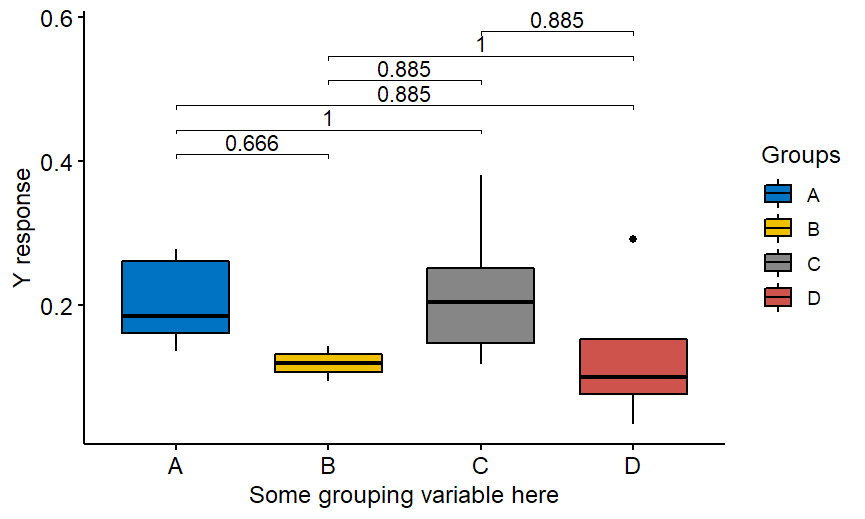
Zauważ, że powyższa metoda dopasowania nie jest już metodą Bonferroniego. Jest to Holm, domyślna metoda zaimplementowana w R. Miłej zabawy!
