
Paarweise Post-hoc-Vergleiche gehören für Datenwissenschaftler zur täglichen Routine. Wann immer wir ANOVA oder nichtparametrische Tests mit mehr als zwei Gruppen durchführen, ist dies unvermeidlich.
Irgendwann möchten Sie vielleicht eine schöne Visualisierung dessen haben, was signifikant ist und was nicht. Möglicherweise mit p-Werten, die in das Diagramm geschrieben werden. Dann können Sie sich eine selbst geschriebene Funktion ausdenken, die diese Aufgabe erfüllt. Oder Sie finden vielleicht eine ggpubr Paket als die einfache Lösung. Und hier kommt die Falle. Der übliche Code, der als Beispiel angegeben wird, erzeugt keine für Mehrfachvergleiche bereinigten p-Werte! Nehmen wir an, Sie haben eine y-Antwort und 4 Gruppen A, B, C, D und Sie möchten y in den anderen Gruppen mit y in der Referenzgruppe A vergleichen. Dann erhalten Sie einen Code wie diesen:
library(tidyverse)
bibliothek(rstatix)
bibliothek(ggpubr)
bibliothek(ggplot2)
meine_Vergleiche <- list( c("A", "B"), c("A", "C"), c("A", "D"))
ggboxplot(Daten, x = "Gruppierung.Variable", y = "Ihre.Antwort",
palette = "jco",fill="gruppierung.variable",order=c("A", "B", "C", "D"),
legend="right")+
stat_compare_means(comparisons = my_comparisons, label.y = c(0.4, 0.5, 0.6),method="wilcox.test",p.adjust.method = "bonferroni")+
labs(y= "Y-Antwort",x= "Hier eine Gruppierungsvariable",fill="Gruppen")
Und die Grafik, die KEINE bereinigten p-Werte enthält!
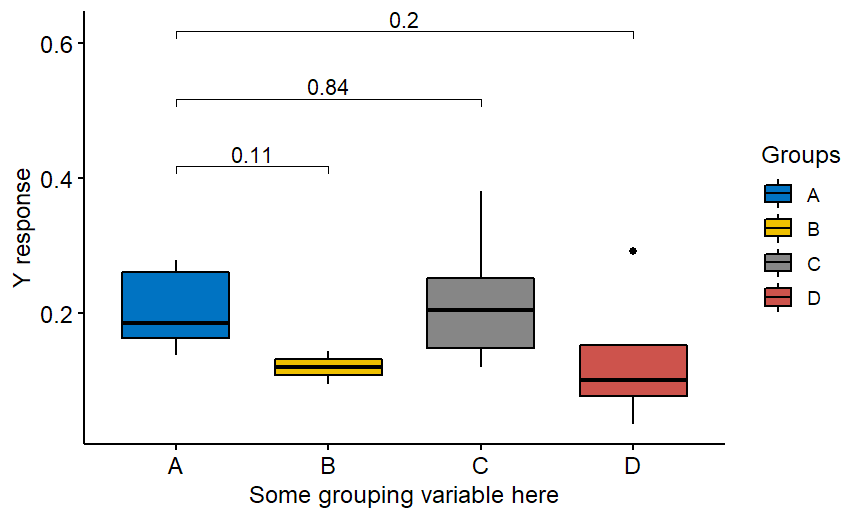
Der richtige Code ist der folgende:
stat.test %
wilcox_test(Ihre.Antwort ~ Gruppierung.Variable,ref.Gruppe = "A") %>%
wert_anpassen() %>%
add_signifikanz("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(Daten, x = "Gruppierung.Variable", y = "Ihre.Antwort",
palette = "jco",fill="gruppierung.variable",order=c("A", "B", "C", "D"),
legend="right")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0.01)+
labs(y= "Y-Antwort",x= "Hier eine Gruppierungsvariable",fill="Gruppen")
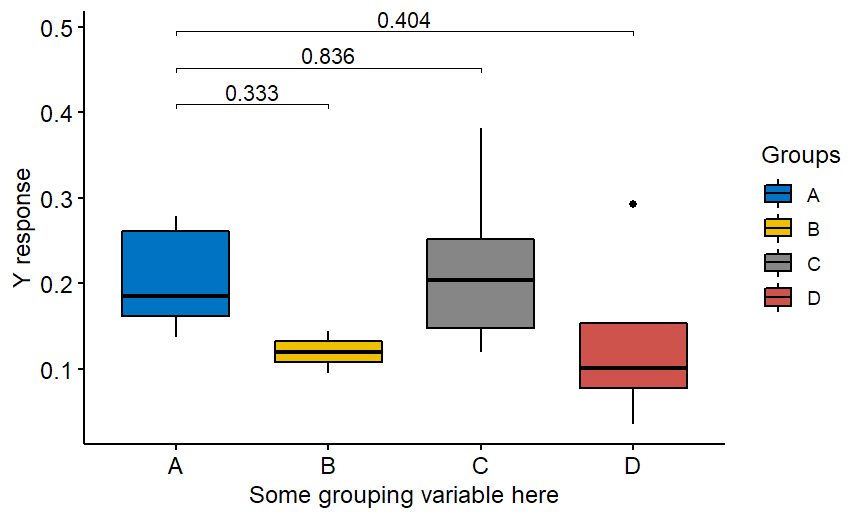
Natürlich ist das obige Beispiel ein nichtparametrischer Wilcoxon-Test, aber das Paket bietet auch t-Tests. Wenn Sie "alles mit allem" vergleichen möchten, nicht nur mit der Gruppe A, dann entfernen Sie einfach ref.group = "A" aus dem Code:
stat.test %
wilcox_test(ihre.antwort ~ gruppierung.variable) %>%
anpassen_wert() %>%
add_signifikant("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(Daten, x = "Gruppierung.Variable", y = "Ihre.Antwort",
palette = "jco",fill="gruppierung.variable",order=c("A", "B", "C", "D"),
legend="right")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0.01)+
labs(y= "Y-Antwort",x= "Hier eine Gruppierungsvariable",fill="Gruppen")Und Sie bekommen:
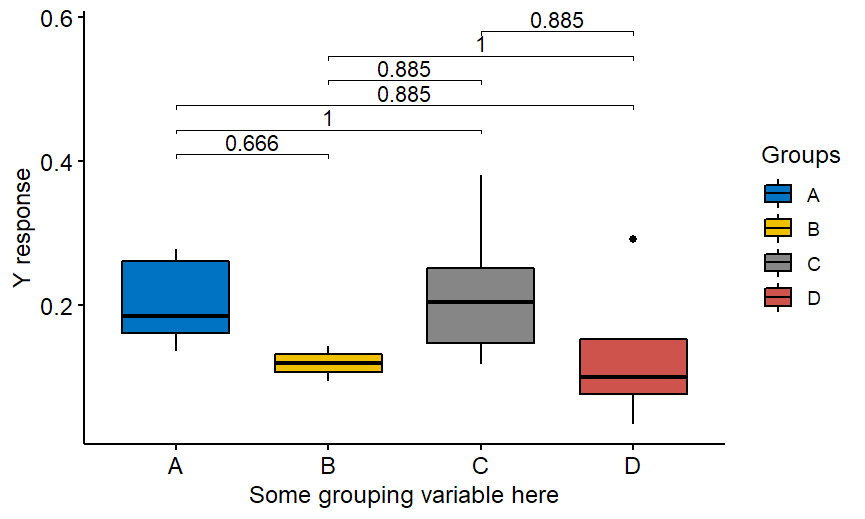
Beachten Sie, dass die obige Anpassungsmethode nicht mehr Bonferroni ist. Es ist Holm, eine in R implementierte Standardmethode. Viel Spaß!
