
Post-hoc paarsgewijze vergelijkingen maken heel vaak deel uit van de dagelijkse routine van de datawetenschapper. Telkens wanneer we ANOVA of niet-parametrische testen doen met meer dan twee groepen is dat onvermijdelijk.
Op een gegeven moment zou je misschien een mooie visualisatie willen hebben van wat significant is en wat niet. Eventueel met p-waarden geschreven op de plot. Dan kun je denken aan een eigen geschreven functie die het werk doet. Of je vindt misschien een ggpubr pakket als de gemakkelijke oplossing. En hier komt de valkuil. De gebruikelijke code die als voorbeeld wordt gegeven produceert geen p-waarden gecorrigeerd voor meervoudige vergelijkingen! Stel dat u een bepaalde y-respons hebt en 4 groepen A, B, C , D en dat u de y in de andere groepen wenst te vergelijken met de y in referentiegroep A. Dan kom je uit op de code zoals deze:
bibliotheek(tidyverse)
bibliotheek(rstatix)
bibliotheek(ggpubr)
bibliotheek(ggplot2)
mijn_vergelijkingen <- lijst( c("A", "B"), c("A", "C"), c("A", "D")
ggboxplot(data, x = "grouping.variable", y = "your.response",
palet = "jco",fill="groepering.variabele",order=c("A", "B", "C", "D"),
legenda="rechts")+
stat_compare_means(vergelijkingen = mijn_vergelijkingen, label.y = c(0.4, 0.5, 0.6),method="wilcox.test",p.adjust.method = "bonferroni")+
labs(y="Y antwoord",x="Een of andere groeperingsvariabele hier",fill="Groepen")
En de grafiek die GEEN aangepaste p-waarden heeft!
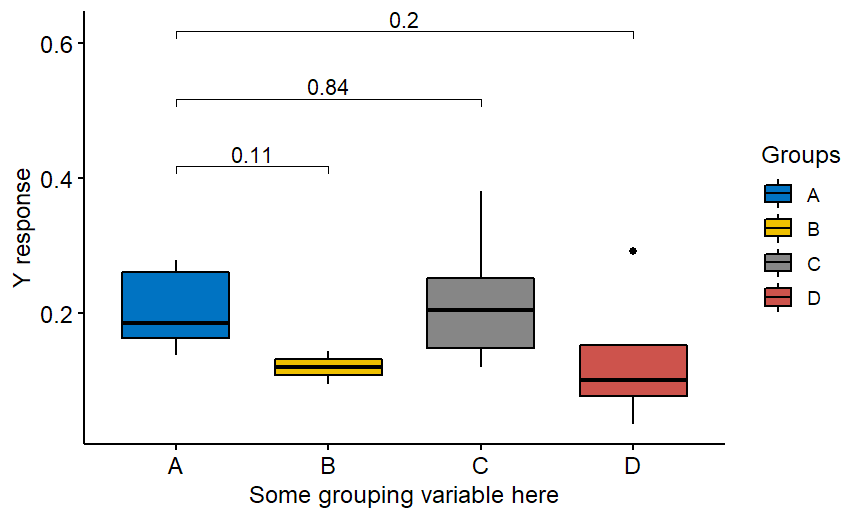
De juiste code is de volgende:
stat.test %
wilcox_test(your.response ~ grouping.variable,ref.group = "A") %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(data, x = "grouping.variable", y = "your.response",
palet = "jco",fill="groepering.variabele",order=c("A", "B", "C", "D"),
legenda="rechts")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0.01)+
labs(y="Y antwoord",x="Een of andere groeperingsvariabele hier",fill="Groepen")
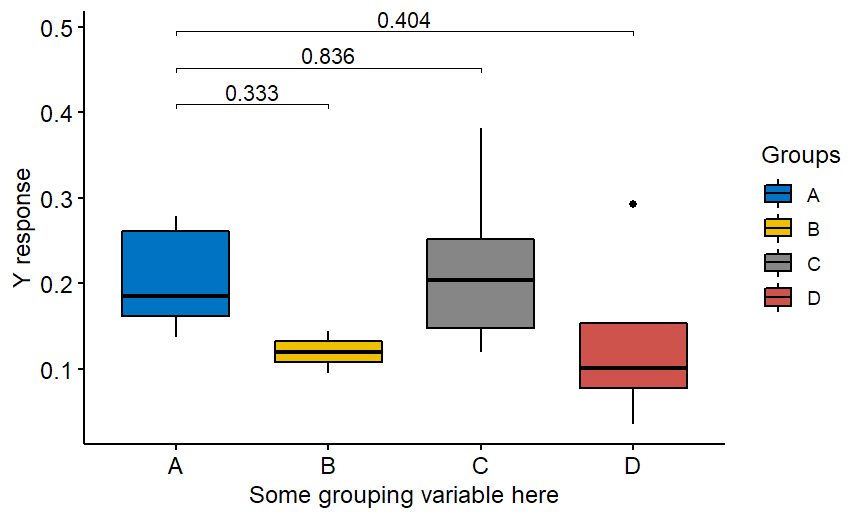
Natuurlijk is bovenstaand een voorbeeld met Wilcoxon niet-parametrische test, maar het pakket biedt ook t-tests. Indien u "alles met alles" wenst te vergelijken, niet alleen met groep A, verwijder dan ref.group = "A" uit de code:
stat.test %
wilcox_test(uw.respons ~ groepering.variabele) %>%
adjust_pvalue() %>%
add_significance("p.adj")
stat.test
stat.test % add_y_position()
ggboxplot(data, x = "grouping.variable", y = "your.response",
palet = "jco",fill="groepering.variabele",order=c("A", "B", "C", "D"),
legenda="rechts")+stat_pvalue_manual(stat.test, label = "p.adj",tip.length = 0.01)+
labs(y="Y antwoord",x="Een of andere groeperingsvariabele hier",fill="Groepen")En je krijgt:
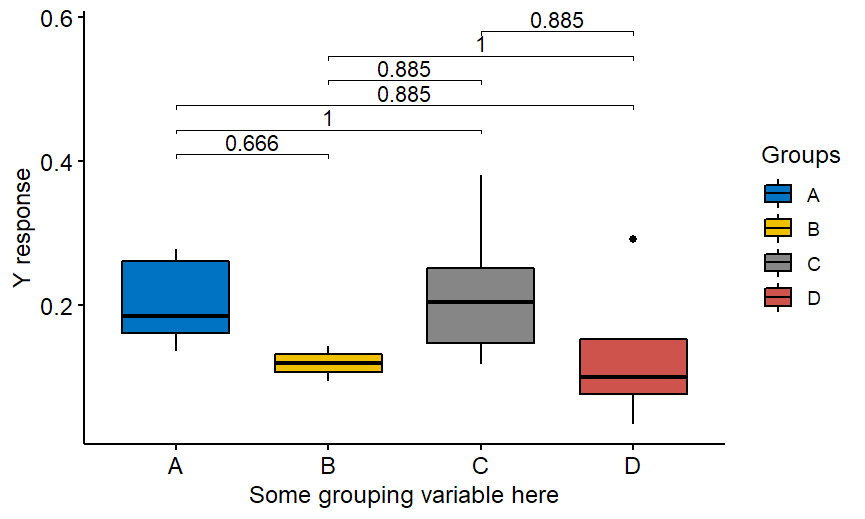
Merk op dat de aanpassingsmethode hierboven niet meer Bonferroni is. Het is Holm, een standaard methode geïmplementeerd in R. Veel plezier!
